De laatste spijker in de doodskist van de menselijke evolutie?
De eerste schattingen van de genetische gelijkenis tussen mens en chimpansee werden gemaakt in de jaren 70. DNA van de twee soorten werd gemengd en herhaaldelijk verhit en afgekoeld in een reageerbuis. Door licht door de buis te laten schijnen, werd de mate van DNA-uitlijning geschat aan de hand van hoe troebel de oplossing werd bij verschillende temperaturen. Dit leverde ons de eerste beweringen op van een “98 tot 99%”-identiteit.1 John Ahlquist, die we interviewden in Creation magazine, was een van de belangrijkste onderzoekers in dat project. Het probleem met deze methode, realiseerde hij zich, is dat deze alleen het DNA meet dat zou passen. Er zouden enorme gebieden met verschillend DNA kunnen bestaan, en ze zouden het niet hebben kunnen zien.2
De meeste evolutionisten accepteerden het cijfer van “98 tot 99%” zonder vragen te stellen, en sindsdien is het op vrijwel alle mediaplatforms verspreid. De enige serieuze uitdaging kwam van de creationistische gemeenschap,3 met slechts af en toe een bekentenis vanuit de evolutionaire hoek. In één daarvan erkende de auteur de “mythe van 1%” en zei dat het werkelijke aantal veel lager ligt.4
Lange tijd leek alles in een cirkeltje te draaien. Toen schreef evolutionair bioloog Dr. Richard Buggs in 2018 een blogpost waarin hij concludeerde dat minder dan 85% van het genoom van mens en chimpansee letter voor letter overeenkwam. 5 Dit was gebaseerd op een artikel dat hij op het punt stond te publiceren met een van zijn pas afgestudeerde promovendi, Josiah Seaman. 6 Dat artikel leek een gelijkenis van 96,6% aan te geven, maar dat gebeurt pas nadat de centromeren, telomeren, alle variaties in het aantal kopieën, ongeveer 300.000 kleine inserties en deleties (goed voor ongeveer twee miljoen letters in elk genoom) en een extra percentage DNA dat zich verzette tegen uitlijning, zijn weggelaten.7 Ja, mensen en chimpansees delen veel DNA, maar als je ook de delen meerekent die ze niet delen, daalt het percentage identiteit aanzienlijk, tot ongeveer 80%.
Nieuw onderzoek met volledige data
Al dit eerdere werk (van drs. Ahlquist, Tomkins, Buggs, enz.) was gebaseerd op onvolledige sequentiebepaling. Pas in de zomer van 2023. 8 beschikten we over een volledig gesequenced menselijk genoom, laat staan over een hoogwaardig chimpanseegenoom. Sterker nog, de vroege versies van het chimpanseegenoom waren zelfs geassembleerd op een basis van het menselijk genoom, waardoor ze automatisch meer op mensen leken. Een belangrijke nieuwe studie heeft echter elk genoom van de mensaap tot een zeer hoge standaard voltooid. 9 Net als het nieuwste menselijke genoom zijn ze (bijna) compleet, van begin tot eind. Oudere versies vertoonden grote hiaten, hadden vervalste centromeergegevens en waren zeer problematisch rond de talrijke repetitieve regio’s. Deze ’telomeer-tot-telomeer’-versies losten deze problemen op.
De assemblage van deze genomen was geen triviale zaak. Zelfs na al dat werk moesten ze een filterprotocol ontwikkelen dat bepaalde DNA-varianten afwees, en moesten ze handmatig meerdere secties selecteren die zich niet ‘gedroegen’.
De uitlijning was nog moeilijker. Ze identificeerden 175 inversies groter dan 10.000 nucleotiden, één chromosoomfusie en één grote translocatie. 10 Ze zagen ook 632 inversies die uniek waren voor slechts één soort.
Analyse van de data
De gegevens over het ‘percentageverschil’ worden gerapporteerd in de aanvullende informatie van Yoo et al. (figuur 1).
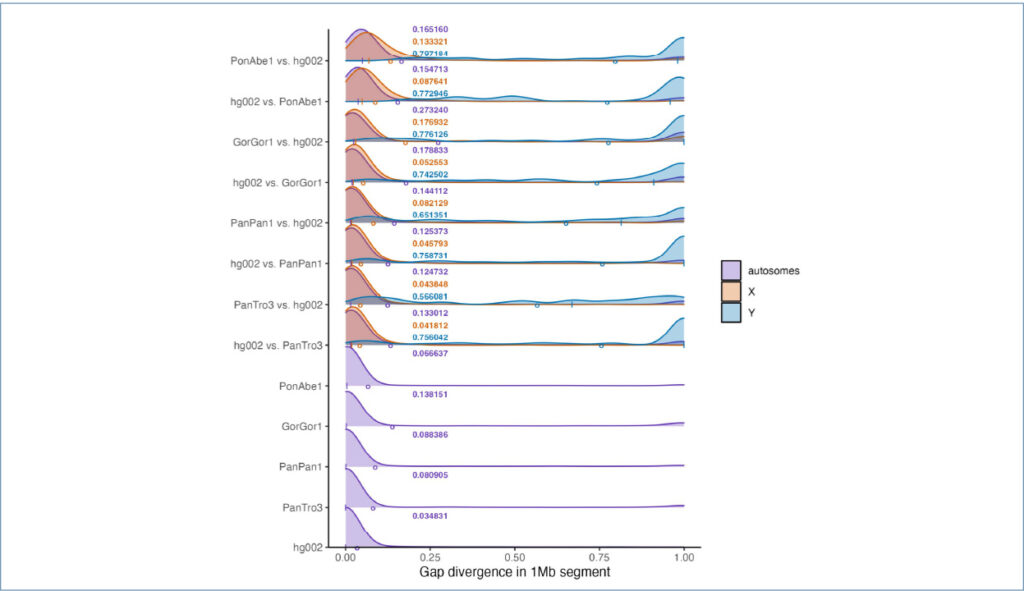
Figuur 1: Het procentuele verschil veroorzaakt door uitlijningsverschillen tussen mens en mensapen. Elke uitlijning werd verdeeld in segmenten van 1 miljoen bp en het procentuele verschil veroorzaakt door de verschillen in dat segment werd berekend. De curven representeren histogram-achtige (bijv. telling) gegevens en elke curve is genormaliseerd naar de maximumwaarde (zodat de pieken allemaal dezelfde hoogte hebben). Het gemiddelde voor elke curve wordt aangegeven door de korte verticale lijnen en de waarden worden gerapporteerd in de kolom met getallen. De curven zijn vaak behoorlijk scheef, waardoor een ‘gemiddelde’ moeilijker te zien is, dus de auteurs rapporteerden ook de mediaan (middelste waarde, cirkels). hg002 = mens, PanTro3 = chimpansee, PanPan3 = bonobo, GorGor1 = gorilla, PonAbe1 = orang-oetan.
Ze gaven geen foutenbalken, alleen het gemiddelde voor elk genoompaar. Ze verdeelden de gegevens ook over de autosomen en de X- en Y-chromosomen. De blauwe bulten rechts in figuur 1 vertegenwoordigen enorme gaten in de Y-chromosoomuitlijning. De paarse bulten onderaan vertegenwoordigen de verschillen die zijn gevonden tussen de twee genoomkopieën binnen hetzelfde individu. Ik heb hun gegevens verwerkt om een complete schatting te krijgen van de divergentie tussen de soorten (figuur 2).
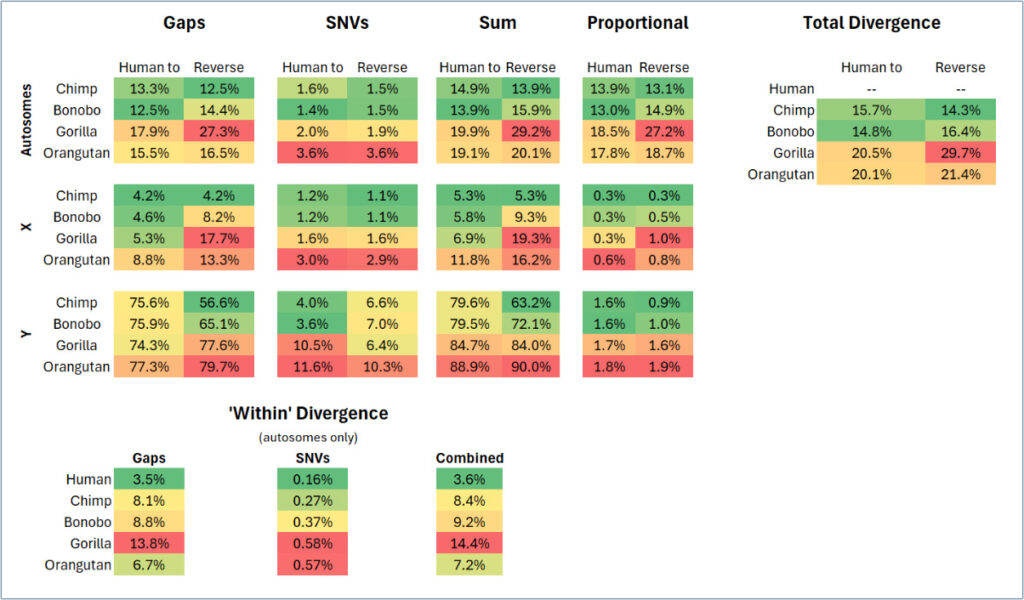
Figuur 2: Berekeningen van genoomgelijkenis. De gegevens voor gap- en SNV-divergentie voor de autosomen en X- en Y-chromosomen werden getabelleerd. De verschillen werden opgeteld en vervolgens gecombineerd met behulp van de proportionele lengtes van de drie chromosoomtypen in de verschillende soorten. De mens-aap- en de omgekeerde berekeningen zijn niet identiek vanwege de aanwezigheid of afwezigheid (afhankelijk van de kijkrichting) van veel uitlijningsgaten.
Daarmee kon een staafdiagram worden gemaakt waarin de verschillen binnen en tussen soorten werden weergegeven (figuur 3).
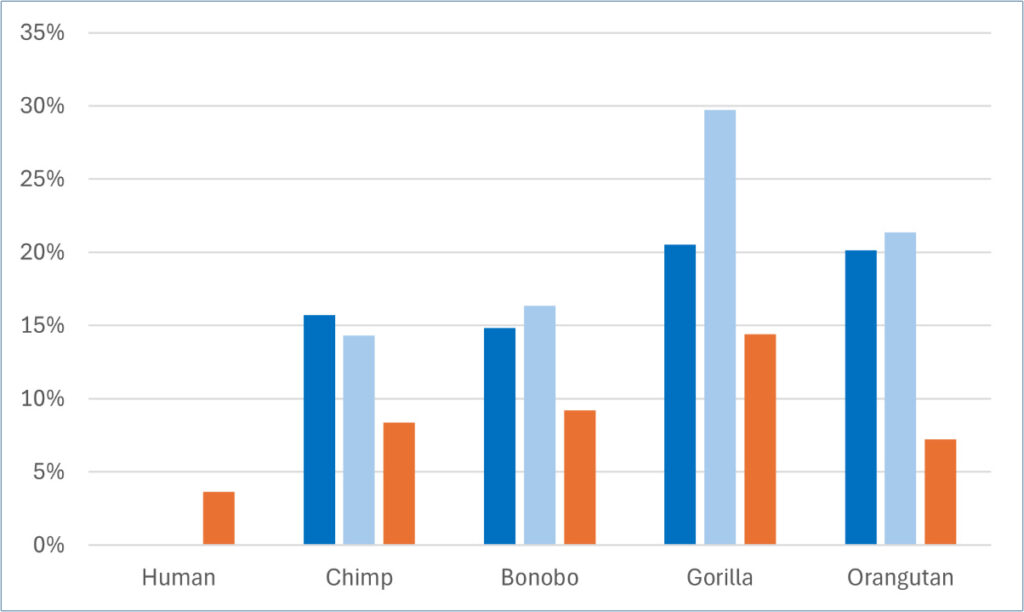
Figuur 3: Definitieve gegevenssamenvatting. Oranje: verschil binnen de soort. Donkerblauw: van mens tot aap. Lichtblauw: van aap tot mens.
99%? Niet eens bijna!
Dit is een belangrijke zaak. Ten eerste hebben veel commentatoren beweerd dat dit bewijst dat het genoom van de mens en de chimpansee slechts voor ongeveer 85% identiek is.11, 12, 13 Ze hebben helemaal gelijk, maar dat zou je niet weten aan de hand van de belangrijkste krantenkoppen. Je kunt de informatie ook niet gemakkelijk vinden in de gepubliceerde publicatie. In plaats daarvan moet je meer dan 100 pagina’s aan gedetailleerde aanvullende informatie doorspitten om de relevante informatie te vinden.
Ten tweede zijn creationistische onderzoekers zoals Dr. Jeffrey Tomkins in het gelijk gesteld. Hoewel eerdere studies werden belemmerd door een gebrek aan betrouwbare sequentiegegevens, laten de nieuwste genomen zien hoe verschillend we zijn van alle apen.
Ten derde zijn er echter enorme stukken sequentie die vrijwel identiek zijn tussen mensen en chimpansees. Ik vond er willekeurig een die zich uitstrekte over enkele honderdduizenden basen en bijna 99% identiek was. 14
Ten vierde zullen onze tegenstanders tot hun laatste adem misleiden en vertroebelen. Een voorbeeld: in een recente video beweerde commentator Gutsick Gibbon dat ik het met haar eens ben over de gelijkenis tussen mens en chimpansee. 15 Nee, hoewel ik het met haar eens ben dat Tomkins een mens is en fouten heeft gemaakt (ik heb ook al op flagrante fouten in haar werk gewezen en ook op diverse fouten in mijn eigen werk 16), ben ik het niet met haar eens dat mensen en chimpansees sterk op elkaar lijken.
Ten vijfde had God ons voor 99,9999% identiek aan chimpansees kunnen scheppen, of voor 50%, of zelfs minder. Het Bijbelse scheppingsmodel doet hier geen eerdere uitspraken over. We zouden een grote gelijkenis moeten verwachten vanwege de duidelijke structurele, gedragsmatige, fysiologische en nutritionele overeenkomsten tussen ons. Maar hoeveel? Niemand kan het weten! Merk op dat de evolutionaire gemeenschap ook niet in staat was om een schatting te maken voordat de aantallen werden berekend. Ze hebben geen voorafgaande toezegging gedaan over welke apensoort meer op ons zou lijken. Het debat over welke soort genetisch gezien meer op ons leek, duurde voort tot de jaren 80. Veel mensen wilden dat de orang-oetans zouden winnen en verzetten zich tegen het idee dat chimpansees onze evolutionaire neven waren. Sommige paleontologen pleitten nog in 2009 voor het primaat van de orang-oetans, hoewel ze toen een zeer kleine minderheid vormden. 17
De vier getallen
Tot slot zijn er vier dingen die we moeten weten:
- Wat is de procentuele overeenkomst tussen de delen die overeenkomen?
- Wat is de procentuele overeenkomst als je de delen meerekent die dat niet doen?
- Hoeveel mutaties moeten er in de loop van de evolutie hebben plaatsgevonden om deze verschillen te verklaren?
- Hoe lang zou het duren om nieuwe mutaties functioneel in het genoom te integreren?
Het antwoord op de eerste vraag is nu bekend: ongeveer 98%. Het antwoord op de tweede vraag is nu ook bekend: ongeveer 85%. Die derde vraag is nu waar het debat over zou moeten gaan, en de vierde vraag is misschien wel de grootste achilleshiel voor evolutietheoretici.
Het evolutionaire model hier verschilt van het onze doordat het ‘eenzijdig’ is. Zij accepteren graag hoge niveaus van overeenkomst, maar er is een klif aan de andere kant van de discussie. Als we te verschillend zijn, kunnen ze de verschillen in hun 6,5 miljoen jaar niet verklaren. Ze hebben wel de mogelijkheid om de tijd terug te brengen tot de meest recente gemeenschappelijke voorouder, maar zelfs dat vermogen is beperkt. Een paar jaar geleden beweerden sommige wetenschappers dat het 13 miljoen jaar zou zijn. 18 Sommigen wilden de tijd nog verder terugschuiven, maar de paleontologen wilden daar niets van weten, omdat ze dan de vroege apen bij de dinosauriërs zouden moeten plaatsen. Ze zitten vast. Het verschil moet klein zijn. Punt.
Hoeveel ‘verschil’ kunnen ze verklaren?
Gegeven een evolutionaire lopende band van nieuwe mutaties die binnenkomen, oude mutaties die door selectie en drift worden verwijderd, en echt oude mutaties die ‘gefixeerd’ worden (d.w.z. 100%), verwachten ze dat de mutatiesnelheid de fixatiesnelheid benadert.
De berekeningen werken als volgt uit:
- Gegeven een haploïde mutatiesnelheid μ, is het aantal nieuwe mutaties per generatie simpelweg 2Nμ, waarbij N de populatiegrootte is.19
- Voor een nieuwe neutrale mutatie is de kans op fixatie evenredig met de frequentie ervan in de populatie. Omdat er 2N kopieën van het genoom in de populatie zijn, en omdat een nieuwe mutatie per definitie begint in één kopie van één chromosoom, is de frequentie van die mutatie 1/(2N). 20 De fixatiesnelheid (r) zou evenredig zijn met het aantal mutaties dat verschijnt (2Nμ).
- Dus, r = 2Nμ/2N = μ
Als de mutatiesnelheid 100 per individu per generatie bedraagt, komt dat neer op 50 mutaties per haploïd genoom per generatie. Ze verwachten dus dat het menselijk genoom 50 vaste verschillen per generatie opbouwt. 21 Over een periode van 6,5 miljoen jaar (~300.000 generaties) verwachten ze 15 miljoen verschillen tussen elke soort en onze gemeenschappelijke voorouder, oftewel ongeveer 30 miljoen verschillen tussen ons en hen vandaag de dag.
30 miljoen verschillen / 3 miljard letters = 1%
DIT is waarom ze al die jaren dat cijfer van 1% hebben aangehaald, en daarom hebben ze zich verzet tegen alles wat anders was. Als het getal veel groter is, werkt het niet in hun voordeel. Wanneer het verschil groter wordt, vallen hun modellen in duigen. Het is gewoon te moeilijk om zoveel verschillen te verklaren, zelfs met hun ‘miljoenen jaren’-mentaliteit.
Maar zelfs een verschil van 15% zou nog steeds verklaard kunnen worden als grote inserties en deleties plotselinge veranderingen veroorzaken. Bedenk dat het Y-chromosoom van een chimpansee slechts half zo lang is als dat van een menselijke Y. Komt dat neer op 30 miljoen verschillen, of op één? Als bijvoorbeeld één deletie de heterochromatische arm van het Y-chromosoom van een chimpansee zou uitwissen, zou er onmiddellijk een verschil van 0,5% tussen onze twee genomen verschijnen. Welke andere grote veranderingen zouden door zulke dingen teweeggebracht kunnen worden?
Maar we hebben het niet over veranderingen in ‘junk-DNA’. Meervoudig functionele genen bevinden zich in de niet-uitgelijnde gebieden. Hoewel ongeveer 99% van de menselijke genen ook bij andere soorten voorkomt, vonden Yoo et al. 185 genfamilies die uniek zijn voor mensen en ongeveer 1.400 tot 2.000 verschillen in het aantal genkopieën tussen de soorten. 22 Dat klopt, veel van de gedupliceerde gebieden hebben te maken met zeer repetitief, niet-coderend DNA, maar deze gebieden blijken steeds vaker functioneel te zijn, zoals wij en anderen al vaak hebben aangegeven. 23 Er zijn ook volledig functionele genen in deze gebieden, met name genen die te maken hebben met de hersenfunctie. 24 Gemiddeld bestaat ongeveer 55% van elk genoom uit repetitieve elementen (LINE’s, SINE’s, LTR’s, etc.). 25 Ook deze blijken functies te hebben en kunnen daarom bij geen enkele vergelijking worden genegeerd.
Gegeven de vele miljoenen puntmutaties en tienduizenden inserties, deleties, inversies en duplicaties, kunnen ze dit verklaren in een evolutionaire context? Ze kunnen sommige ervan verklaren in hun modellen, maar die modellen zijn vaak vrij simplistisch (zoals de bovenstaande vergelijkingen). Willekeurige paring is een cruciale aanname, maar die klopt nooit, en niet-willekeurige paring vertraagt alleen maar de snelheid waarmee nieuwe varianten zich verspreiden. Er zijn ook vragen over de bevolkingsgroei en hoe deze alle berekeningen beïnvloedt. Aangezien de menselijke bevolking zich heeft uitgebreid (sinds de zondvloed of sinds de uitvinding van de landbouw, kies maar), hebben zich in de afgelopen 10.000 jaar in de evolutionaire tijdlijn GEEN genetische varianten vastgelegd in het menselijk genoom. Hoe beïnvloedt ‘geen evolutie gedurende 10.000 jaar’ de evolutionaire voorspelling?
Maar de vierde vraag hierboven is misschien wel de meest fundamenteel belangrijke vraag in de evolutie. Waarom? Omdat evolutie nieuwe genen nodig heeft om te ontstaan en te activeren. Mensen en chimpansees verschillen niet alleen op nucleotideniveau. Onze genen worden niet op dezelfde manier gebruikt en onze hersenen hebben heel andere bedradingspaden. Die veranderingen zouden niet zomaar moeten ontstaan. Nee, ze zouden moeten ontstaan, zich verspreiden en het oorspronkelijke gen op die plek vervangen, en zich vervolgens integreren in de toch al complexe regulatieprocessen die er zijn.
This is a massive problem even for the “1%” crowd. Now that we know the human and chimpanzee genomes are more than 10 times more different than they thought, the problem of evolution only becomes that much more difficult. This is one of the greatest scientific discoveries that supports the biblical creation model. It does not mean, however, that evolutionists will never be able to explain what we see. It does mean, though, that they will be scrambling for cover. The ‘fact’ that they have been trumpeting from the rooftops since the 1970s turns out to be no fact at all. The real difference is NOT 1%. No, it is 15x greater.
- See the discussion in Carter, R.W., Reassessing human–chimpanzee genetic similarity, J. Creation 38(1):93–103, 2024. ↩︎
- This was one of the things that broke the Darwinian mindset and caused him to turn to the Bible. See Wieland, M., Convert to creation: Margaret Wieland interviews bird expert and former renowned evolutionist Dr Jon Ahlquist, Creation 40(3):36–39, 2018. ↩︎
- Tomkins, J. and Bergman, J., Genomic monkey business—estimates of nearly identical human–chimp DNA similarity re-evaluated using omitted data, J. Creation 26(1):94–100, 2012. See ref 1 for a fuller list of citations. ↩︎
- Cohen, J., Relative differences: the myth of 1%, Science 316(5833):1836, 2007. ↩︎
- Buggs, R., How similar are human and chimpanzee genomes? Richardbuggs.com, 14 Jul 2018 ↩︎
- Seaman, J. and Buggs, R., FluentDNA: nucleotide visualization of whole genomes, annotations, and alignments, Frontiers in Genetics 11:292, 2020 ↩︎
- Kuderna, L., Complete ape genomes offer a close-up view of human evolution, Nature 641(8062):313–314, 2025 ↩︎
- Rhie, A. et al., The complete sequence of a human Y chromosome, Nature 621(7978):344–354, 2023 ↩︎
- Yoo, D. et al., Complete sequencing of ape genomes, Nature 641(8062):401–418, 2025 ↩︎
- Yoo et al., ref. 7, supplementary information, p. 101 ↩︎
- Luskin, C, Letter to the Smithsonian: Correct your signage on human-chimp genetic similarity! evolutionnews.org, 27 May 2025 ↩︎
- Buggs, R., How much of a human genome is identical to a chimpanzee genome? richardbuggs.com, 6 May 2025 ↩︎
- Tomkins, J.P., Chimp genome markedly different from human, icr.org, 29 May 2025 ↩︎
- Carter, R.W., Reassessing human–chimpanzee genetic similarity, J. Creation 38(1):93–103, 2024 ↩︎
- Gutsick Gibbon, I killed this creationist argument, youtube.com, 28 May 2025 ↩︎
- Carter, R., James 3 vs the anticreationists, biblicalgenetics.com, 16 Jan 2024; youtube.com/watch?v=FIY7FTTFZyg ↩︎
- Grehan, J.R. and Schwartz, J.H., Evolution of the second orangutan: phylogeny and biogeography of hominid origins, J. Biogeogr. 36(10):1823–1844, 2009 ↩︎
- Venn, O. et al., Strong male bias drives germline mutation in chimpanzees, Science 344(6189):1272–1275, 2014 ↩︎
- The formula includes a “2” because the genome is diploid. The mutation rate is usually given as the haploid mutation rate, for historical reasons ↩︎
- Again, a 2 is in the denominator because there are two copies of the genome per individual in diploid species ↩︎
- Given random mating, which never happens, and a stationary population size, which is clearly not true for humans. Without these assumptions the evolutionary model cannot deliver even a 1% difference ↩︎
- Yoo et al., ref. 7, supplementary information pp. 66–67 ↩︎
- See our ‘Vestigial’ Organs Questions and Answers ↩︎
- Kuderna, L., Complete ape genomes offer a close-up view of human evolution, Nature 641(8062):313–314, 2025 ↩︎
- Yoo et al., ref. 7, supplementary information p. 78 ↩︎


Nog geen reacties